Autoři
Iva Dolinová, Roman Špánek, Alena Ševců, Lukáš Dvořák
Klíčová slova
chlorované uhlovodíky – biologická dekontaminace – podzemní voda – real-time PCR – sekvenace druhé generace – molekulárně-genetické analýzy
Chlorované uhlovodíky (ClU) patří mezi nejčastější kontaminanty životního prostředí. Posun od mechanických způsobů jako je odtěžení či chemických (oxidativní i reduktivní) forem čištění směrem ke kombinovaným, tj. chemicko-biologickým nebo čistě biologickým postupům vytvořil prostor pro uplatnění molekulární genetiky i v této oblasti. Pomocí molekulárně-genetických metod je možné jak detailně monitorovat průběh mikrobiální degradace, tak predikovat její vývoj a podle toho nastavovat vhodné parametry sanačního procesu.
V předkládaném článku jsou shrnuty jednotlivé metody a vysvětleny možnosti jejich použití, výhody i limitace. Dále jsou uvedeny případové studie, ve kterých jsou hodnoceny konkrétní lokality a situace z pohledu molekulární genetiky. Použití uvedených metod však není omezeno pouze na kontaminované lokality či podzemní vody, ale tyto metody lze efektivně aplikovat na kterékoli procesy čištění vod, respektive na všechny vzorky, kde jsou přítomny mikroorganismy.
1. Úvod
Chlorované uhlovodíky (ClU) patří mezi nejrozšířenější kontaminanty životního prostředí. V 2. polovině minulého století byl vyráběn především perchloretylen (PCE), který byl hojně používán k chemickému čištění oděvů či jako rozpouštědlo a průmyslové odmašťovadlo. Vlivem nedbalého nakládání s těmito látkami došlo k jejich únikům a rozsáhlým kontaminacím životního prostředí, tedy i podzemních vod, kde se ClU staly jedním z nejčastějších kontaminantů.
Pro odstraňování ClU je možné využít jak biologických, tak chemických metod či jejich vzájemných kombinací, které se v současnosti jeví jako nejefektivnější. Kombinace biologických a chemických metod se začaly hojněji využívat až v 90. letech minulého století. Rozvoj metod molekulární genetiky, a to zejména pokročilých sekvenačních metod, navíc umožnil rozsáhlé studium mechanismů biologické degradace ClU. Prostřednictvím metod molekulární genetiky lze monitorovat jak průběh vlastní biodegradační reakce, tak i predikovat její vývoj, čímž může být celý sanační zásah efektivně řízen a optimalizován.1.1. Biodegradace chlorovaných uhlovodíků
Kromě původní kontaminace způsobené perchloretylenem (PCE) a trichloretylenem (TCE) je na kontaminovaných lokalitách v podzemních vodách přítomna také tzv. „sekundární“ kontaminace. Ta je způsobena přirozeně vznikajícími meziprodukty rozkladu PCE a TCE, tj. dichloretylenem (cis-DCE) a vinylchloridem (VC), které jsou navíc toxičtější než původní kontaminanty [1, 2]. Biodegradace PCE a TCE za anaerobních podmínek probíhá přes méně chlorované uhlovodíky cis-, trans-, 1,2- a 1,1- dichloretyleny a dále přes vinylchlorid až na etylen.
Do biodegradace je zapojeno široké spektrum bakterií s různými dehalogenačními schopnostmi. Přítomnost a aktivita těchto bakteriálních konsorcií je determinována vlastnostmi daného prostředí. Schopnost bakterií degradovat ClU není omezena pouze na anaerobní prostředí, ale může probíhat také v aerobních podmínkách [3]. V anaerobním prostředí je však biodegradace (anaerobní respirace) mnohem častější, přičemž ClU jsou využity jako akceptory elektronů. Při aerobních podmínkách jsou ClU naopak využity jako donory elektronů. Za aerobních i anaerobních podmínek může také docházet ke kometabolickému rozkladu kontaminantů, přičemž pro správný průběh rozkladu je nutná přítomnost dalšího organického substrátu, např. derivátu kyseliny octové, etanu, etenu, fenolu či toluenu [1, 4, 5].
1.2. Anaerobní biodegradace
Reduktivní dehalogenace je hlavním degradačním pochodem v anaerobním prostředí [6]. Kromě anaerobních podmínek je nutná přítomnost donoru elektronů – vodíku, který vzniká fermentací organických látek. Jako akceptor elektronů slouží přítomné ClU, přičemž energie uvolněná při dehalogenační reakci je využita pro růst bakterií. Rychlost reduktivní dehalogenace je ovlivněna jak biogeochemickými procesy, tak i množstvím dostupného vodíku [7]. Akumulace toxických meziproduktů, konkrétně cis-DCE a VC, může být způsobena jak částečnou dechlorací, tak sníženou rychlostí dechlorace, eventuálně jejich vzájemnou kombinací [3]. Jediným doposud známým mikroorganismem, který je schopný degradovat PCE až na etan/eten pomocí anaerobní reduktivní dehalogenace, je bakteriální rod Dehalococcoides [8]. Dále bylo popsáno množství druhů, které degradují vždy pouze určitý ClU, například Desulfitobacterium spp., Dehalobacter spp. a Sulfurospirillum multivorans [9].

1.3. Aerobní biodegradace
Další možnou cestou dekontaminace ClU je jejich aerobní biodegradace. Během ní jsou ClU využity jako donor elektronů, přičemž dostatečné jsou i velmi nízké koncentrace kyslíku [10]. Aerobní biodegradace ClU je popsána pro TCE , cis-DCE [1] a VC [11]. Bakteriálních druhů schopných aerobní degradace VC je v porovnání s anaerobní biodegradací mnohem více. Jedná se např. o Mycobacterium sp. [12], Nocardioides sp. [13], Ochrobactrum sp. [14], Pseudomonas aeruginosa [15] či Ralstonia sp [16]. Při aerobní biodegradaci se uplatňují především enzymy mono- a dioxygenázy. Aerobní biodegradace však není v praxi příliš často využívána, jelikož v jejím průběhu může docházet k nežádoucímu těkání jednotlivých ClU do okolního prostředí.
1.4. Kometabolická biodegradace
Kometabolická cesta biodegradace ClU probíhá jak za aerobních, tak za anaerobních podmínek. Při tomto procesu biodegradace jsou ClU degradovány „náhodně“. Tzn., že do metabolických drah příslušných bakterií, např. metanogenních nebo síru a železo redukujících, vstupují ClU společně s jinými substráty, aniž by je bakterie degradovaly s jakýmkoliv ziskem energie či uhlíku [17]. Tyto bakterie využívají jako primární substrát jiné látky než ClU (např. toluen, katechol, metan, etanol a jiné). Z tohoto důvodu jsou ClU při kometabolickém rozkladu degradovány enzymy s jinou substrátovou specifitou. Pokud poklesne koncentrace primárního substrátu pod určitou hladinu, dojde k zastavení celého kometabolického procesu [18]. Enzymy zapojené do kometabolické biodegradace jsou převážně mono- a dioxygenázy, konkrétně například katechol 1,2-dioxygenáza, toluen dioxygenáza a metan monooxygenáza [19]. Kometabolické biodegradace ClU se může účastnit mnoho bakteriálních druhů, např. Pseudomonas sp. [20], Comamonas testosteroni [21], Mycobacterium vaccae [22], Rhodococcus sp. [23] a další.
2. Metodická část – přehled nejběžnějších molekulárně-genetických metod
V současné době jsou velmi intenzivně sledovány změny ve složení autochtonní mikroflóry podílející se na biodegradaci nejrůznějších kontaminantů z podzemních vod. V případě ClU se jedná především o proces reduktivní dehalogenace za anaerobních podmínek [24]. S rozvojem molekulárně-genetických metod dochází k hlubšímu poznání nejen mikrobiálních společenstev, ale i enzymů zapojených do jednotlivých biodegradačních drah. Detekci změny hladin konkrétních bakteriálních druhů či specifických biodegradačních enzymů lze využít nejen při analýzách kontaminovaných vod, ale i například při testování aktivity mikroorganismů aktivovaného kalu apod.
2.1. Polymerázová řetězová reakce
Základní molekulárně-genetickou metodou, která umožňuje získat přehled o procesech probíhajících na kontaminované lokalitě, je polymerázová řetězová reakce (PCR). Jedná se o metodu, na jejímž principu a modifikaci je postavena většina dalších molekulárně-genetických analýz. Na obr. 1a je uveden princip této metody. V prvním kroku dojde k denaturaci molekul DNA pomocí vysoké teploty. Druhý krok slouží k navázání specifických primerů, což jsou krátké sekvence DNA, které se specificky váží na konkrétní místa genomu. Teplota, při které probíhá druhý krok, se odvíjí od jejich sekvence. V třetím kroku dochází k syntéze vlastního vlákna, a tedy ke vzniku nové kopie testované DNA. Výše popsané kroky se cyklicky opakují tak, aby došlo k dostatečnému namnožení testované sekvence a bylo možné signál vizualizovat (obr. 1b).
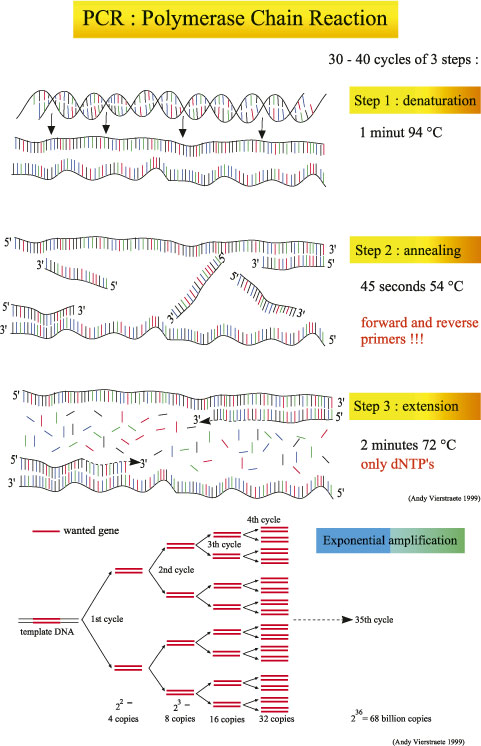
Obr. 1. a, b) Princip polymerázové řetězové reakce [25]
Doba jednotlivých kroků je závislá na použitých chemických činidlech, proto údaje uvedené na obrázku jsou pouze orientační. Konečným krokem zviditelnění signálu je v tomto případě gelová elektroforéza, jejíž výsledek je uveden na obr. 1c. Podle přítomnosti signálu v gelu lze reakci jednoduše vyhodnotit – bakterie nebo specifický enzym je, či není přítomen.
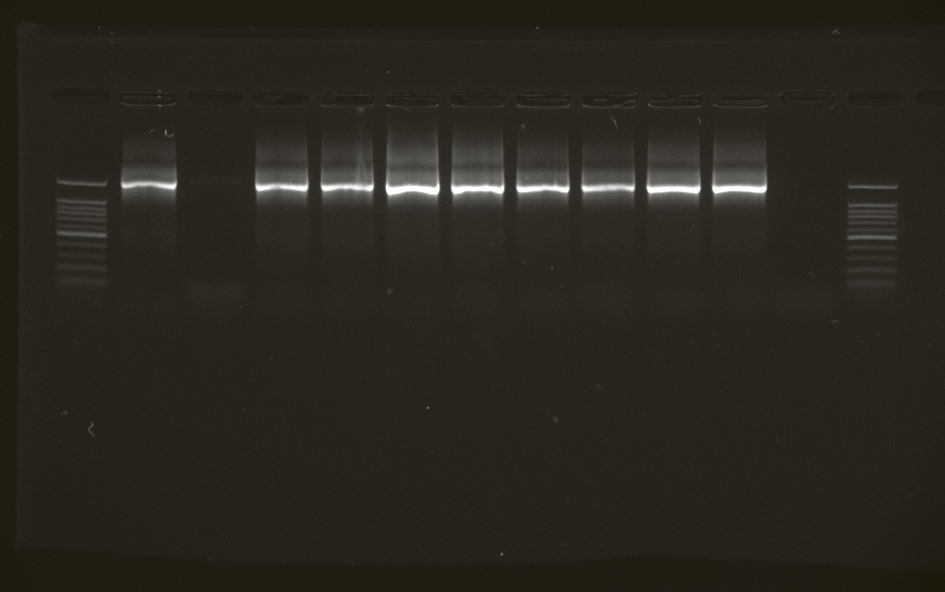
Obr. 1 c) výsledek PCR reakce na gelové elektroforéze
2.2. Real-time PCR analýza
Jedná se o pokročilou formu PCR založené na detekci fluorescenčního signálu. Ten je vyzářen buď pomocí sond, nebo fluorescenční barvy (např. SYBR Green). Princip vyzáření fluorescenčního signálu je patrný z obr. 2a, b.
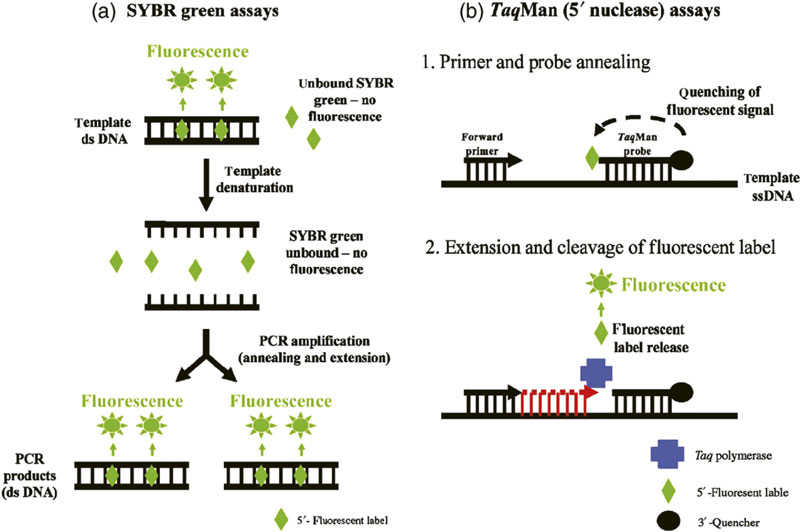
Obr. 2. a, b) Princip real-time PCR detekce
Měření fluorescenčního signálu po skončení každého cyklu PCR reakce. Intenzita fluorescence následně slouží k výpočtu relativní nebo absolutní kvantity (obr. 2c).

c) výsledek amplifikace cílového úseku [26]
Relativní kvantifikace umožňuje zjišťování trendů, tedy růst nebo pokles signálu vůči původnímu stavu. Při absolutní kvantifikaci dochází k přesnému určení počtu kopií, proto lze hodnotit jak trendy, tak jednotlivé vzorky navzájem.
V případě biodegradace ClU (aerobní i anaerobní cestou) lze pomocí real-time PCR sledovat jak pokles či nárůst signálu pro geny enzymů predikujících aktivně probíhající biodegradační proces (např. pceA, vcrA, bvcA, monooxygenázy, hydrolázy), tak i jednotlivé bakteriální rody podílející se aktivně na biodegradaci ClU (např. Dehalococcoides spp., Dehalobacter spp., Pseudomonas spp.).2.3 Sangerovo sekvenování
Sangerovo sekvenování je verifikačním nástrojem sloužícím ke kontrole např. specifičnosti zvolených primerů nebo k přesnému taxonomickému zařazení čistých bakteriálních kultur získaných kultivačními metodami. Metodika je založená opět na principu PCR reakce a využívá možnosti fluorescenčního značení dideoxynukleotidů (ddNTP), za kterými již není možné pokračovat v syntéze nově vznikajícího řetězce DNA (obr. 3).
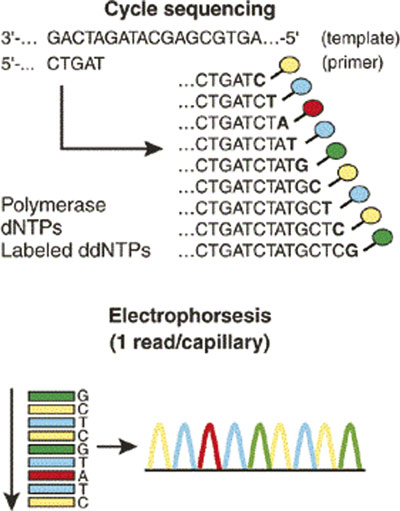
Obr. 3. Princip Sangerova sekvenování [27]
2.4. Next Generation Sequencing (NGS) analýza
NGS analýza je relativně nový přístup molekulární genetiky, který nachází uplatnění v širokém spektru analýz. V případě environmentálních vzorků lze NGS metodou detailně analyzovat směsné vzorky bakteriálních komunit. Tato metoda, jako jediná, analyzuje složení komplexní bakteriální komunity bez nutnosti klonování sledovaných úseků DNA do vektorů a následné kultivace v buňce. Analyzovány a porovnávány mohou být například bakteriální komunity z různých hloubek, z různých kontaminovaných lokalit či jiných vzorků. Jediným omezením NGS metody je vstupní koncentrace izolované DNA, která musí být vyšší než 2 ng·µl-1. Pokud je koncentrace izolované DNA nižší, mohou být získané výsledky odlišné od reálného stavu.
Metoda NGS též umožňuje sekvenovat celé bakteriální genomy, a to jak v případě směsných vzorků (metagenom), tak i komplexní genomy izolátů bakteriálních kmenů. Touto metodou lze například zjistit degradační potenciál bakteriální komunity či konkrétního bakteriálního kmene.

Obr. 4. Princip NGS sekvenace na přístroji Ion Torrent [28]
3. Případové studie pro jednotlivé molekulárně-genetické metody
V této kapitole jsou představeny jednotlivé molekulárně-genetické metody vhodné pro analýzy biodegradace ClU, včetně konkrétních výsledků.
3.1. PCR detekce vybraných markerů
Dvě různé lokality kontaminované ClU byly hodnoceny pomocí vybraných markerů a metody PCR. V tabulce 1 je uveden výsledek monitoringu bakterií schopných dehalogenace (Dehalococcoides spp., Dehalobacter spp., Sulfurospirillium spp.) a specifických genů pro expresi enzymů, které se účastní degradace VC (vcrA, bvcA). Jediná kvantifikace, kterou PCR analýza umožňuje je +/-, tedy přítomen/nepřítomen.

Tabulka 1. Ilustrační výsledky PCR reakce ze dvou lokalit kontaminovaných ClU. Sanační zásah (aplikace syrovátky) byl proveden po prvním a druhém odběru. Vysvětlivky: – marker nepřítomen, + marker přítomen, +/- marker přítomen na hranici detekce
Lokalita 1 vykázala po zásaku syrovátky (organický substrát) pozitivní trend ve vývoji specifického bakteriálního osídlení, přičemž při posledním odběru byla detekována přítomnost všech sledovaných markerů, a tedy úspěšně probíhající proces biodegradace. Proto lze konstatovat, že aplikace syrovátky měla pozitivní vliv na vývoj biodegradačních procesů. Lokalita 2 vykazovala dlouhodobou stabilitu výskytu specifických biodegradérů a probíhající biodegradaci. Z výsledků PCR reakce nelze zhodnotit, zda měla aplikace syrovátky další podpůrný vliv na specifickou mikroflóru.
3.2. Real-time PCR detekce vybraných markerů
Real-time PCR reakce umožňuje přesnou kvantifikaci vývoje jednotlivých sledovaných markerů. Sanační zásahy lze proto monitorovat a vyhodnocovat jak v krátkodobém, tak dlouhodobém časovém horizontu.
Obr. 5 ilustruje vliv aplikace glycerolové fáze (substrátu) v čase „0“ na specifickou bakteriální mikroflóru v sanačním vrtu s přítomností ClU.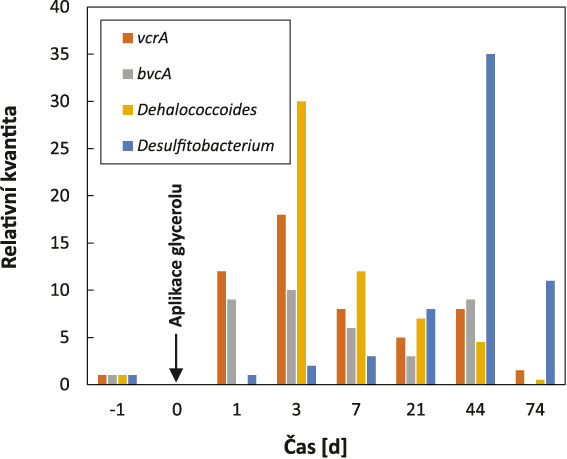
Obr. 5. Vliv aplikace substrátu (glycerolové fáze) na vývoj relativní kvantifikace vybraných molekulárně-genetických markerů reflektujících biodegradační procesy
Po aplikaci substrátu do vrtu došlo k nárůstu relativní četnosti u všech sledovaných markerů. Jak je patrné z tohoto obrázku, již jeden den po aplikaci byl pozorován vzestup hladin obou sledovaných specifických enzymů pro degradaci VC, tj. vcrA i bvcA. Třetí den od aplikace došlo také k nárůstu relativní četnosti markeru pro Dehalococcoides spp. Bylo tedy identifikováno zvýšení všech markerů spojených s biodegradací ClU. Od třetího dne, kdy jednotlivé markery dosáhly nejvyšších hodnot relativní kvantity, byl dále pozorován jejich pokles, a to na hodnoty přibližně stejné jako před aplikací. Naopak pozitivní vliv na vývoj dehalogenační mikroflóry ve vrtu a přispěla k rychlejší biodegradaci ClU.
3.3. NGS analýza autochtonní mikroflóry
Cílem NGS analýzy bylo v tomto případě porovnat diverzitu bakterií v podzemní vodě kontaminované ClU s diverzitou bakterií narostlých na nanovlákenných nosičích umístěných ve stejné vodě. Dále byly analyzovány změny ve složení bakteriální komunity na této lokalitě mezi dvěma odběry, kdy byla do vrtu aplikována syrovátka jako substrát pro podporu biodegradačních procesů. NGS analýza tedy měla za cíl reflektovat případné změny ve složení autochtonní mikroflóry ovlivněné touto aplikací.
Z výsledků analýz uvedených na obr. 6 je patrné, že bakteriální diverzita se na úrovni kmene, v případě prvního odběru, podobala diverzitě bakterií detekované na nanovlákenném nosiči. Rozdíly mezi 1. a 2. odběrem, tedy před a po aplikaci syrovátky, byly však již znatelné (obr. 6).
Obr. 6. Výsledky NGS sekvenování na úrovni kmene; porovnání složení bakteriální komunity přítomné ve vzorku podzemní vody a nanovlákenného nosiče a vzorků vody mezi 1. a 2. odběrem (před a po aplikaci syrovátky)
Majoritní výskyt ve vzorku podzemní vody vykázala čeleď Methylophilaceae, zatímco na nanovlákenném nosiči byla tato čeleď zastoupena je velmi raritně. Ostatní skupiny se víceméně shodovaly a jednotlivé rozdíly byly způsobené především vlastnostmi sekvenování, než skutečnými rozdíly v diverzitě. Byl však pozorován významný posun ve složení bakteriální komunity mezi 1. a 2. odběrem. Došlo k potlačení kmene Proteobacteria a naopak k proliferaci kmene Firmicutes. Tato skupina je přímo spojována s degradací ClU a zároveň je přítomna v syrovátce. Rozdílné složení mezi oběma odběry je patrné i na úrovni čeledí (obr. 7b).

Obr. 7. Porovnání dat získaných pomocí NGS sekvenování na úrovni: a) kmene, b) čeledi s frekvencí zastoupení nad 5 %
Byly pozorovány tři majoritní čeledi již dříve publikované v souvislosti s výskytem na lokalitách kontaminovaných chlorovanými etyleny (Ruminococcaceae, Moraxellaceae a Lachnospiracea). Změna může být vysvětlena aplikací malého množství syrovátky (0,2 m3) do tohoto vrtu, navíc s nízkou rychlostí proudění podzemní vody. Bakterie kmene Firmicutes jsou v syrovátce významně zastoupeny, tudíž ovlivnily i 2. odběr, respektive následné výsledky NGS analýzy.
Souhrnně lze konstatovat, že NGS analýzy poskytují komplexní obraz o dané lokalitě z hlediska mikrobiálního osídlení. Většina detekovaných čeledí byla již dříve popsána v souvislosti s výskytem na kontaminované lokalitě nebo i přímo s biodegradací ClU.4. Závěr
V předloženém článku byly shrnuty dostupné metody molekulární genetiky a představeny jejich hlavní benefity a limitace. Jednotlivé možnosti použití daných molekulárně-genetických metod byly doloženy konkrétními případovými studiemi, respektive výsledky získanými při monitoringu reálných kontaminovaných lokalit. Nejprve byla představena základní metoda molekulární genetiky, tj. PCR reakce, která umožňuje prostou detekci jednotlivých sledovaných markerů. Dále byla popsána pokročilejší metoda real-time PCR. Ta již umožňuje kvantifikaci získané informace a dovoluje proto vyvozovat případné trendy ve vývojích. NGS analýza, jako nejpokročilejší metoda molekulární genetiky, umožňuje proniknout až do samé podstaty jednotlivých dějů či detailního složení bakteriální mikroflóry přítomné ve sledovaném vzorku.
Všechny metody popisované v tomto příspěvku lze využít pro studium libovolných biologických vzorků, tedy např. při monitoringu mikrobiálních pochodů probíhajících nejen v kontaminovaných podzemních vodách či půdách, ale také při detailním sledování a hodnocení procesů na čistírnách odpadních vod.Poděkování
Tento příspěvek vznikl za finanční podpory Grantové agentury České republiky, v rámci projektu No. 14-32432S a v rámci projektu LM2015073 NanoEnviCz, podpořeného MŠMT.
Literatura/References
[1] T. E. Mattes, A. K. Alexander, N. V. Coleman, Aerobic biodegradation of the chloroethenes: pathways, enzymes, ecology, and evolution, FEMS Microbiol. Rev. 34 (2010) 445–475.[2] I. Nijenhuis, M. Nikolausz, A. Köth, T. Felföldi, H. Weiss, J. Drangmeister, J. Großmann, M. Kästner, H.-H. Richnow, Assessment of the natural attenuation of chlorinated ethenes in an anaerobic contaminated aquifer in the Bitterfeld/Wolfen area using stable isotope techniques, microcosm studies and molecular biomarkers, Chemosphere. 67 (2007) 300–311.
[3] G. Imfeld, H. Pieper, N. Shani, P. Rossi, M. Nikolausz, I. Nijenhuis, H. Paschke, H. Weiss, H. H. Richnow, Characterization of groundwater microbial communities, dechlorinating bacteria, and in situ biodegradation of chloroethenes along a vertical gradient, Water. Air. Soil Pollut. 221 (2011) 107–122.
[4] T. Kruse, J. Maillard, L. Goodwin, T. Woyke, H. Teshima, D. Bruce, C. Detter, R. Tapia, C. Han, M. Huntemann, Complete genome sequence of Dehalobacter restrictus PER-K23 T, Stand. Genomic Sci. 8 (2013) 375.
[5] P. J. McMurdie, S. F. Behrens, J. A. Müller, J. Göke, K. M. Ritalahti, R. Wagner, E. Goltsman, A. Lapidus, S. Holmes, F. E. Löffler, Localized plasticity in the streamlined genomes of vinyl chloride respiring Dehalococcoides, PLoS Genet. 5 (2009) e1000714.
[6] M. M. Häggblom, D. E. Fennell, Y.-B. Ahn, B. Ravit, L. J. Kerkhof, anaerobic dehalogenation of halogenated organic compounds: novel strategies for bioremediation of contaminated sedimentsof contaminated sedimentsof contaminated sedimentsof contaminated sediments, in: Soil Water Pollut. Monit. Prot. Remediat., Springer, 2006: pp. 505–521.
[7] J. C. Chambon, P. L. Bjerg, C. Scheutz, J. Bælum, R. Jakobsen, P. J. Binning, Review of reactive kinetic models describing reductive dechlorination of chlorinated ethenes in soil and groundwater, Biotechnol. Bioeng. 110 (2013) 1–23.
[8] M. Duhamel, E. A. Edwards, Microbial composition of chlorinated ethene-degrading cultures dominated by Dehalococcoides, FEMS Microbiol. Ecol. 58 (2006) 538–549.
[9] B.-E. Jugder, H. Ertan, M. Lee, M. Manefield, C. P. Marquis, Reductive dehalogenases come of age in biological destruction of organohalides, Trends Biotechnol. 33 (2015) 595–610.
[10] T. M. Vogel, C. S. Criddle, P. L. McCarty, ES&T critical reviews: transformations of halogenated aliphatic compounds, Environ. Sci. Technol. 21 (1987) 722–736.
[11] M. Morkin, J. Devlin, J. Barker, B. Butler, In situ sequential treatment of a mixed contaminant plume, J. Contam. Hydrol. 45 (2000) 283–302.
[12] B. Z. Fathepure, V. K. Elango, H. Singh, M. A. Bruner, Bioaugmentation potential of a vinyl chloride-assimilating Mycobacterium sp., isolated from a chloroethene-contaminated aquifer, FEMS Microbiol. Lett. 248 (2005) 227–234.
[13] M. M. Chartrand, A. Waller, T. E. Mattes, M. Elsner, G. Lacrampe-Couloume, J. M. Gossett, E. A. Edwards, B. Sherwood Lollar, Carbon isotopic fractionation during aerobic vinyl chloride degradation, Environ. Sci. Technol. 39 (2005) 1064–1070.
[14] A. S. Danko, M. Luo, C. E. Bagwell, R. L. Brigmon, D. L. Freedman, Involvement of linear plasmids in aerobic biodegradation of vinyl chloride, Appl. Environ. Microbiol. 70 (2004) 6092–6097.
[15] M. F. Verce, R. L. Ulrich, D. L. Freedman, Transition from cometabolic to growth-linked biodegradation of vinyl chloride by a Pseudomonas sp. isolated on ethene, Environ. Sci. Technol. 35 (2001) 4242–4251.
[16] V. K. Elango, A. S. Liggenstoffer, B. Z. Fathepure, Biodegradation of vinyl chloride and cis-dichloroethene by a Ralstonia sp. strain TRW-1, Appl. Microbiol. Biotechnol. 72 (2006) 1270–1275.
[17] D. J. Arp, C. M. Yeager, M. R. Hyman, Molecular and cellular fundamentals of aerobic cometabolism of trichloroethylene, Biodegradation. 12 (2001) 81–103.
[18] L. Alvarez-Cohen, G. E. Speitel Jr, Kinetics of aerobic cometabolism of chlorinated solvents, Biodegradation. 12 (2001) 105–126.
[19] Y. Li, B. Li, C.-P. Wang, J.-Z. Fan, H.-W. Sun, Aerobic degradation of trichloroethylene by co-metabolism using phenol and gasoline as growth substrates, Int. J. Mol. Sci. 15 (2014) 9134–9148.
[20] H. Futamata, S. Harayama, K. Watanabe, Diversity in kinetics of trichloroethylene-degrading activities exhibited by phenol-degrading bacteria, Appl. Microbiol. Biotechnol. 55 (2001) 248–253.
[21] H. Futamata, S. Harayama, K. Watanabe, Group-specific monitoring of phenol hydroxylase genes for a functional assessment of phenol-stimulated trichloroethylene bioremediation, Appl. Environ. Microbiol. 67 (2001) 4671–4677.
[22] L. P. Wackett, S. R. Householder, Toxicity of trichloroethylene to Pseudomonas putida F1 is mediated by toluene dioxygenase, Appl. Environ. Microbiol. 55 (1989) 2723–2725.
[23] O. Suttinun, R. Müller, E. Luepromchai, Cometabolic degradation of trichloroethene by Rhodococcus sp. strain L4 immobilized on plant materials rich in essential oils, Appl. Environ. Microbiol. 76 (2010) 4684–4690.
[24] F. Maphosa, W. M. de Vos, H. Smidt, Exploiting the ecogenomics toolbox for environmental diagnostics of organohalide-respiring bacteria, Trends Biotechnol. 28 (2010) 308–316.
[25] Vierstracte, A., 1999. http://users.ugent.be/~avierstr/principles/pcr.html, ověřeno k 6. 9. 2016.
[26] C. J. Smith, A. M. Osborn, Advantages and limitations of quantitative PCR (Q-PCR)-based approaches in microbial ecology, FEMS Microbiol. Ecol. 67 (2009) 6–20.
[27] I. U. Leong, J. R. Skinner, D. R. Love, Application of massively parallel sequencing in the clinical diagnostic testing of inherited cardiac conditions, Med. Sci. 2 (2014) 98–126.
[28] Genomics, http://www.genomics.cn/en/navigation/show_navigation?nid=2640, ověřeno k 6. 9. 2016. Mgr. Iva Dolinová 1, 2) (autor pro korespondenci)
Ing. Roman Špánek, Ph.D. 1)
RNDr. Alena Ševců, Ph.D. 1)
Ing. Mgr. Lukáš Dvořák, Ph.D. 1)


